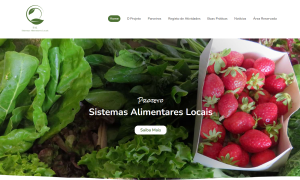
TERRA FUTURA
Uma Agricultura mais sustentável, competitiva
e inovadora, emissora e recetora de conhecimento.
Uma Agricultura do País, da Europa e do Mundo
de todas e de todos...
Ambiente e
Recursos Naturais
Iniciativas emblemáticas:
«Mitigação das alterações climáticas»
«Adaptação às alterações climáticas»
- Conservação e fertilidade do solo
- Gestão dos recursos hídricos
- Aumentar a resiliência dos ecossistemas
agrícolas espécies e habitats aos efeitos
das alterações climáticas - ...
Sistemas Agrários
Iniciativas emblemáticas:
«Agricultura circular»
«Territórios sustentáveis»
«Revitalização das zonas rurais»
«Agricultura 4.0»
«Promoção dos produtos agroalimentares
portugueses»
Sanidade Vegetal
Iniciativa emblemática:
«UMA SÓ SAÚDE»
- Reduzir a incidência de doenças das plantas
- Impulsionar a adaptação da produção
vegetal às ameaças emergentes - ...
Sistemas Florestais
Iniciativa emblemática:
«Territórios sustentáveis»
- Serviços de ecossistema: desenvolver e
valorizar os serviços ecológicos, promover
a biodiversidade, a apicultura e a silvopastorícia. - ...
Saúde Animal
Iniciativa emblemática:
«UMA SÓ SAÚDE»
- Reduzir a incidência de doenças dos animais
com impacto na saúde e bem-estar da
população humana e no ambiente - Reduzir a emergência à resistência a
antimicrobianos - ...
Produção Animal
Iniciativas emblemáticas:
«Agricultura circular»
«Territórios sustentáveis»
«Revitalização das zonas rurais»
«Agricultura 4.0»
«Promoção dos produtos agroalimentares
portugueses»
Segurança Alimentar
Iniciativas emblemáticas:
«Alimentação sustentável»
«Agricultura circular»
«Agricultura 4.0»
«Promoção dos produtos agroalimentares
portugueses»
Recursos Genéticos
Eixo estratégico:
«Valorização e gestão sustentável
dos recursos naturais e genéticos»
- promover a conservação, melhoramento e
valorização dos recursos genéticos de
natureza animal e vegetal - variedades regionais e as raças autóctones, com avaliação de variedades e raças
selecionadas em modelos de produção
comercial